摘要
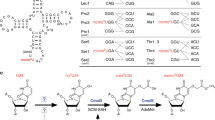
许多转移rna的转录后修饰在翻译中起着至关重要的作用。2-甲基硫代- n6 -异五烯基腺苷(ms2i6A)修饰发生在反密码子36位腺嘌呤的转运rna的37位(A37),有助于促进A:U密码子-反密码子碱基的有效配对,并防止近同源物的非预期碱基配对,从而提高翻译保真度1,2,3,4。ms2i6A修饰是由自由基s -腺苷蛋氨酸(SAM)甲基硫转移酶MiaB安装到异戊烯腺苷(i6A)上的。作为一种自由基SAM蛋白,MiaB含有一个[Fe4S4]RS簇,用于SAM的还原裂解,形成一个5′-脱氧腺苷基5′-自由基,负责去除底物的C2氢5。MiaB还含有一个辅助的[Fe4S4]aux簇,该簇参与了硫向i6A37的C2转移6,7,8,9。这种转移是如何发生的在很大程度上是未知的。本文介绍了均匀拟杆菌中MiaB的几种结构。这些结构符合两步机制,其中一个SAM分子首先用于甲基化辅助性簇的桥接微硫离子。在第二步中,第二个SAM分子被切割成一个5′-脱氧腺苷基5′-自由基,它从底物中提取C2氢,但只有在C2从sp2再杂交到sp3之后。这项工作促进了我们对酶如何功能化与硫的惰性碳氢键的理解。
这是订阅内容的预览,可通过您的机构访问
访问选项
访问Nature和其他54种Nature组合期刊
获取自然+,我们最具价值的在线订阅
99€/ 30天
随时取消
中国客户的订阅信息
我们有一个专门的网站为我们的中国客户。请登录naturechina.com订阅本刊。
购买这篇文章
- 在Springer Link上购买
- 即时访问全文PDF
价格可能会在结账时计算当地税收




类似的内容被其他人浏览
数据可用性
本文报道的晶体结构的原子座标和结构因子已被沉积到PDB中,编号为7MJZ(含五硫桥的天然结构)、7MJY(含SAH和13-mer RNA的结构)、7MJV(含SAM和17-mer RNA的结构)、7MJX(含5 -dAH+Met和13-mer RNA的结构)和7MJW(含预甲基化BuMiaB和5 -dAH+Met和13-mer RNA的结构)。
参考文献
Connolly, D. M.和Winkler, M. E.在大肠杆菌K-12中,miaA基因、2-甲基硫代- n6 -(δ 2-异戊烯基)-腺苷tRNA修饰和自发诱变之间的遗传和生理关系。[j] .中华微生物学杂志,2001,11(2):444 - 444。
Connolly, D. M. & Winkler, M. E.大肠杆菌K-12 miaA的结构和由miaA插入突变引起的突变表型的表征。[j] .中华微生物学杂志,2003,17(2):444 - 444。
Esberg, B., Leung, h . c .。E., Tsui, h . c .。T., Björk, G. R. & Winkler, M. E.:鼠伤寒沙门氏菌和大肠杆菌tRNA中参与异戊烯基化A37衍生物甲基硫代化的miaB基因鉴定。[j] .中国生物医学工程学报,2003,19(2):444 - 444。
Urbonavicius, J, Qian, Q, Durand, J. M. B, Hagervall, T. G. & Björk, G. R.改善阅读框维持是几种tRNA修饰的共同功能。[j] .中国生物医学工程学报,2009,32(2):444 - 444。
自由基s -腺苷-l-蛋氨酸(SAM) tRNA甲基硫转移酶MiaB的机制研究。博士论文,宾夕法尼亚州立大学,(2016)。
兰德格拉夫,b.j.,阿奇纳斯,a.j.,李,k.h。s -腺苷甲硫氨酸甲基硫转移酶RimO和MiaB中间甲基载体的鉴定。j。化学。社会科学学报,135,15404 - 15416(2013)。
Zhang, B.等。自由基s -腺苷蛋氨酸甲基硫转移酶MiaB催化的第一步产生具有[3Fe-4S]0样辅助簇的中间体。j。化学。社会学报,142,1911-1924(2020)。
Forouhar, F.等。两个Fe-S簇通过自由基- sam甲基硫转移酶催化硫插入。Nat,化学。生物学报,9,33 - 338(2013)。
Hernández, h.l.等。MiaB是一种双功能自由基- s -腺苷蛋氨酸酶,参与tRNA的硫代化和甲基化,包含两个必需的[4Fe-4S]簇。生物化学46,5140-5147(2007)。
Reiter, V.等。CDK5抑制因子CDK5RAP1是一种作用于细胞核和线粒体RNA的甲基硫转移酶。中国生物医学工程学报,2012,32(2):444 - 444。
魏凤英等。cdk5rap1介导的线粒体trna的2-甲基硫修饰控制蛋白质翻译并有助于小鼠和人类的肌病。中国生物医学工程学报,2015,33(2):428-442。
Adami, R. & Bottai, D. s -腺苷蛋氨酸tRNA修饰:前/新参与者的意外/未预料的影响。Int。生物。科学通报,16(2),2023 - 2023(2020)。
Dhaven, R. & Tsai, l - h。CDK5的十年。中华生物医学杂志,2001,27(2):749-759。
等人。tRNA生物合成2-甲基硫代- n6 -苏酰基氨基腺苷的真核和原核甲基硫代转移酶鉴定。生物。化学,28,28425-28433(2010)。
安东,b.p.等人。枯草芽孢杆菌YmcB和YqeV tRNA甲基硫转移酶的功能表征中国生物医学工程学报,2014,33(2):481 - 481。
Landgraf, b.j., McCarthy, e.l. & Booker, s.j.自由基s -腺苷蛋氨酸酶在人类健康和疾病中的作用。为基础。生物化学学报,35(5):487 - 487(2016)。
安东,b.p.等人。RimO是一种类似miab的酶,可以甲基硫酸化大肠杆菌中普遍保守的核糖体蛋白S12的Asp88残基。自然科学进展。美国105,1826-1831(2008)。
Landgraf, b.j. & Booker, s.j.自由基SAM甲基硫转移酶RimO催化反应的立体化学过程j。化学。社会科学学报,38(1),2889-2892(2016)。
等人。核糖体蛋白的翻译后修饰:来自Thermotoga maritima的RimO的结构和功能表征,一种自由基s -腺苷蛋氨酸甲基硫转移酶。生物。化学,28,572 - 5801(2010)。
Agris, P. F, Armstrong, D. J, Schäfer, K. P. & Söll, D.转移RNA中超修饰核苷的成熟。核酸学报,2691 - 698(1975)。
Molle, T.等。甲基硫转移酶RimO中s -腺苷甲硫氨酸(SAM)结合Fe-S簇的氧化还原行为,以了解双SAM活性。生物化学55,5798-5808(2016)。
在ruma - RNA -辅因子三元复合物中,一个独特的RNA折叠有助于底物选择性和酶的功能。Cell 120,599 - 611(2005)。
Anantharaman, V., Koonin, E. V. & Aravind, L. TRAM,一个预测的rna结合域,与tRNA尿嘧啶甲基化和腺嘌呤硫代化酶共同存在。《。左197,215-221(2001)。
Chimnaronk, S.等。tRNA反密码子合成n6 -异戊烯腺苷的动力学快照。生物化学,48,5057-5065(2009)。
Anantharaman, V., Koonin, E. V. & Aravind, L. RNA代谢中蛋白质的比较基因组学和进化。核酸,30,1427-1464(2002)。
西村,S.在转移RNA:结构,性质和识别卷1(编辑Schimmel, p.r., Sӧll, D. & Abelson, j.r.)(冷泉港实验室出版社,1979)。
波卡莱托,P.等。MODOMICS: RNA修饰途径的数据库。2017年更新。中国生物医学工程学报,2016,33(2):481 - 481。
琼斯,S.蛋白质- rna相互作用:结构分析和功能分类。蛋白质66,903-911(2007)。
Jones, S., Daley, D. T., Luscombe, N. M., Berman, H. M. & Thornton, J. M.蛋白质- rna相互作用:结构分析。核酸学报,29,943-954(2001)。
Pierrel, F., Douki, T., Fontecave, M.和Atta, M. MiaB蛋白是一种双功能自由基- s -腺苷蛋氨酸酶,参与tRNA的硫代化和甲基化。生物。化学学报,27(2004)。
Grove, T. L, Radle, M. I, Krebs, C. & Booker, S. J. Cfr和RlmN含有一个单一的[4Fe-4S]簇,它指导s -腺苷蛋氨酸的两种不同的反应:SN2置换甲基转移和自由基生成。j。化学。社会学报,133,19586-19589(2011)。
Kim, S., Meehan, T.和Schaefer, H. F., III。从腺嘌呤-尿嘧啶碱基对中提取氢原子。期刊。化学。A . 11, 6806-6812(2007)。
Zierhut, M, Roth, W.和Fischer, I.腺嘌呤中h原子损失的动力学。理论物理。化学。化学。物理学报,6,5178-5183(2004)。
兰茨,n.d.等人。RlmN和AtsB作为自由基SAM蛋白过量产生和表征的模型。方法:中国生物医学工程学报(英文版)。
Sambrook, J., Fritsch, e.f. & Maniatis, T.《分子克隆:实验室手册第二版,第三卷》(冷泉港实验室出版社,1989年)。
McCarthy, E. L. & Booker, S. J.了解大肠杆菌脂酰合成酶催化过程中铁硫簇再生的生化方法。方法:中国生物医学工程学报,2018,37(6):2217 - 2239。
Minor, W., Cymborowski, M., Otwinowski, Z.和Chruszcz, M. HKL-3000:数据约简和结构解的集成-从衍射图像到初始模型在几分钟内。Acta Crystallogr。D 62, 859-866(2006)。
特威利格,t.c.等。迭代模型构建,结构细化和密度修改与PHENIX AutoBuild向导。Acta Crystallogr。D 64, 61-69(2008)。
埃姆斯利,P.,洛坎普,B.,斯科特,W. G.和考坦,K.。Acta Crystallogr。[j] .农业工程学报,2016,38(5)。
Afonine, P. V.等。用phenix.refine实现晶体结构的自动细化。Acta Crystallogr。[j] .生物医学工程学报,2012,32(5):556 - 567。
威廉姆斯,c.j.等人。MolProbity:为改进全原子结构验证提供更多更好的参考数据。中国生物医学工程学报,2016,33(4):444 - 444。
Ashkenazy, H.等。ConSurf 2016:一种估计和可视化大分子进化守恒的改进方法。中国生物医学工程学报,2016,33(2):444 - 444。
致谢
这项工作得到了美国国立卫生研究院(NIH) (GM-122595 to S.J.B.;AI133329到S.C.A.和T.L.G.;GM-127079到C.K.;和GM118393, GM093342和GM094662到S.C.A.),国家科学基金会(MCB-1716686到S.J.B.), Eberly家族杰出科学主席(S.J.B.), Price家族基金会(S.C.A.)和宾夕法尼亚州立大学Huck生命科学研究所(N.H.Y.)。s。j。b。是霍华德·休斯医学研究所的研究员。这项研究使用了先进光子源的资源,先进光子源是美国能源部(DOE)科学办公室用户设施,由阿贡国家实验室根据合同编号。DE-AC02-06CH11357。转基因/CA@APS的使用全部或部分由国家癌症研究所(ACB-12002)和国家普通医学科学研究所(AGM-12006)的联邦资金资助。GM/CA-XSD的Eiger 16M探测器由NIH拨款S10 OD012289资助。LS-CAT第21部门的使用得到了密歇根经济发展公司和密歇根技术三走廊的支持(拨款085P1000817)。这项研究还使用了伯克利结构生物学中心的资源,该中心的部分资源得到了霍华德休斯医学研究所的支持。先进光源是美国能源部科学用户设施办公室根据合同编号。DE-AC02-05CH11231。ALS-ENABLE光束线部分由美国国立卫生研究院、美国国家普通医学科学研究所资助,拨款P30 GM124169。
作者信息
作者及单位
贡献
o.a.e.、t.l.g.、N.H.Y.和S.J.B.制定了研究计划和实验策略。O.A.E.和T.L.G.分离并结晶蛋白质并收集晶体学数据。O.A.E, B.W.和A.J.A.进行了生化实验。O.A.E, t.l.g., N.H.Y, S.C.A, C.K.和S.J.B.分析和解释了晶体学数据。o.a.e.、t.l.g.、N.H.Y.和S.J.B.写了手稿,所有其他作者都对它进行了审查和评论。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益冲突。
额外的信息
同行评议信息《自然》感谢匿名审稿人对本文同行评议的贡献。
施普林格·自然对已出版地图的管辖权要求和机构关系保持中立。
扩展数据图和表
扩展数据图1部MiaB和TmRimO结构。
a, BuMiaB(蓝色)和TmRimO (PDB:4JC0)(灰色)结构的卡通叠加。b、TmRimO的静电表面电位(蓝色为正,红色为负,灰色为中性)。c, BuMiaB和TmRimO的氨基酸序列比对。BuMiaB的整体结构与TmRimO相似,两个独立的RimO分子在324和329 Cαs上的rmsd分别为1.5和1.6 Å(表S1)。在RimO x射线晶体结构中,两个[Fe4S4]簇相距7.3 Å(每个簇中最近的离子),并通过跨越每个簇的独特(非半胱氨酸连接)铁的五硫化物桥连接。在BuMiaB结构中也观察到同样的五硫桥,其中簇间距为6.8 Å(见扩展数据图2a)。
扩展数据图2 RNA结合到所有三个结构域部MiaB。
a, BuMiaB活性位点的卡通表示与跨越两个[Fe4S4]簇的五硫桥。mtase结构域;自由基SAM域,灰色;TRAM区域,绿色。b, BuMiaB的卡通在13-mer RNA底物和5'dAH +Met存在下结晶,并显示13-mer在三个结构域界面的结合(紫色)。c,静电表面电位(蓝色为正电,红色为负电,灰色为中性)表示一个带正电的活性位点区域促进13-mer的结合。d,从CONSURF服务器推断的活性位点区域氨基酸的保守性42。
图3 13-mer结合的ACSL结构比较部MiaB与全长tRNA板式换热器绑定到TmMiaA。
a,与BuMiaB和5 ' -dAH+Met配合物中的13-mer(核苷酸29-41)结构(紫色)与Tm tRNAPhe与MiaA配合物(棕色)的卡通覆盖(PDB ID: 2ZM5)。b, 13-mer与BuMiaB之间形成氢键示意图。
图4 17-mer绑定的ACSL结构比较部MiaB与全长tRNA板式换热器绑定到TmMiaA。
a, 13-mer(紫色)和17-mer(核苷酸27-43)(绿色)结构与BuMiaB + 5 ' -dAH+Met (13-mer)或BuMiaB与SAM (17-mer)配合,与Tm tRNAPhe与MiaA(棕褐色)配合(PDB ID: 2ZM5) (PDB ID: 2ZM5)。b, 17-mer与BuMiaB之间形成的氢键示意图。
扩展数据图5部含有反密码子34和35核苷酸的MiaB。
含有13-mer RNA和5'dAH +Met的BuMiaB结构以粉红色表示,含有17-mer RNA和SAM的BuMiaB结构以褐红色表示。a,活性位点与G34的相互作用。G34常被改造,其基底在深裂带中插入TRAM和RS结构域之间,为改造提供了空间。在BuMiaB与5 ' -dAH+Met和13-mer配合物的结构中,G34的N10位于RS结构域Ser388的h键距离内。2 '和3 '羟基与Arg418的两个氮原子成氢键,这些氮原子来自tram结构域的胍基。在含有SAM和17-mer的BuMiaB结构中,G34的位置不同,该碱基不再与Ser388和Arg418相互作用(扩展数据图3b, 4b)。b,活性位点与A35的相互作用。在含有5 ' -dAH+Met和13-mer的BuMiaB结构中,Asp319的羧酸氧与A35的N6在h键距离内,而Gln28的侧链与A35的2 ' OH在h键距离内。A35碱基在Phe348和i6A37的腺嘌呤环之间π堆叠。A35在sam束缚结构中的位置发生移位,并以G34和Phe348为一侧的π堆积来稳定。Phe348的旋转支持A35在酶活性位点的两种不同取向。c,结构中BuMiaB活性位点i6A37与13-mer和5'dAH +Met结合,表明异戊烯基位于疏水斑块中。结构域及其相关残基的颜色均相同:棕褐色为mtase,灰色为自由基SAM,绿色为TRAM。
扩展数据图6全长tRNA结合到部MiaB。
a,从CONSURF服务器导出的BuMiaB中残基守恒。色码在面板中有说明。b,静电表面电位,表示能稳定tRNAPhe的带正电区域。c, MTTase结构域残基与全长tRNA相互作用的预测模型。
图7 SAM、SAH和5 ' -dAH与部MiaB。
a, SAM(灰色),SAH(海蓝宝石)和5 ' dah +Met(浅紫色)在它们与BuMiaB和RNA底物的配合物中的覆盖(SAM为17-mer, SAH或5 ' -dAH+Met为13-mer)。SAM、SAH和5'dAH的腺嘌呤环与Phe321形成面对面的π堆积相互作用。与两个酪氨酸(177,352)和Phe350的边对面相互作用进一步支持这种堆叠。腺嘌呤环的N3与保守的Arg66形成氢键(如图3a所示),N6与Ile65 (mtase结构域)、Tyr177和Ser353 (RS结构域)的羰基形成3个氢键。SAM、SAH和5'dAH的核糖部分与Arg66、Gln281和Asp319键合。5'dAH +Met结构中的蛋氨酸或SAM(与17-mer RNA结合)和SAH(与13-mer RNA结合)结构中的蛋氨酸部分与[Fe4S4]RS簇的独特铁呈典型双齿结合。b, SAM(灰色)或5 ' dah +Met(浅紫色)与BuMiaB和17-mer RNA (SAM)或13-mer RNA (5 ' -dAH+Met)的复盖。i6A37碱基中含有5'dAH +Met的结构为粉红色,含有SAM的结构为栗色。结构域及其相关残基的所有图都有相同的颜色:mtase为褐色,自由基SAM为灰色,TRAM为绿色,除了5'dAH +Met结构中的Gln215(图a),它是旋转的。
图8 Arg66→Gln取代对部MiaB活动。
a, 25 μM BuMiaB WT(黑色圆圈)或BuMiaB R66Q(红色圆圈)与1mm SAM在不含二亚硫石的情况下形成SAH的时间过程。b-e, 25 μM BuMiaB与1 mM SAM初始孵育30 min后,加入100 μM i6A ACSL RNA,用1 mM二硫代盐引发反应,SAH (b), 5'dAH (c), ms2i6A (d), i6A衰变(e)的形成时间。黑色对应于在17-mer RNA底物存在下BuMiaB WT获得的数据;蓝色对应于在13-mer存在下获得的数据;红色对应于17-mer存在时BuMiaB R66Q的数据。误差条表示三次重复反应的一个标准偏差,中心表示平均值。
图9预甲基化活性位点电子密度的立体表示部在13-mer RNA底物和5'dAH +Met存在下的MiaB。
BuMiaB的结构在预甲基化和非预甲基化状态下复合物的蛋白质或RNA组分没有任何显著变化[预甲基化亚基A和B与非预甲基化亚基A相比,RMSD分别为0.231 Å (Cα = 371个原子)和0.089 Å (Cα = 439个原子);预甲基化亚基A和B与非甲基化亚基B相比,分别为0.092 Å (Cα = 415个原子)和0.248 Å (Cα = 392个原子)。a, N3处的扩展电子密度。灰色网格对应的是i6A 3.5σ等高线的Fo-Fc省略图,绿色网格对应的是i6A细化后3.0σ等高线的Fo-Fc图。b, [Fe3S4]簇中硫原子的扩展电子密度。该网格对应于3.5σ辅助簇的甲基(绿色)附着在硫(灰色)上的Fo-Fc省略图。c,在非预甲基化辅助簇生成的图中,没有观察到扩展密度。该网格对应于辅助簇的硫原子在3.5σ处轮廓的Fo-Fc省略图。所有残基的结构域都有一个共同的颜色主题:棕褐色为mtase,灰色为自由基SAM。
权利和权限
关于本文
引用本文
Esakova, o.a., Grove, t.l., Yennawar, N.H.等。自由基SAM酶MiaB对tRNA甲基硫基化的结构基础。自然,597,566-570(2021)。https://doi.org/10.1038/s41586-021-03904-6
收稿日期:2021年4月5日
录用日期:2021年8月12日
发布日期:2021年9月15日
发行日期:2021年9月23日
DOI: https://doi.org/10.1038/s41586 - 021 - 03904 - 6
这篇文章是由
-
trna衍生片段:肺癌基因调控机制及临床应用
细胞肿瘤学(2024)
-
四醚类脂质合成酶的发现、结构及作用机制
自然(2022)
评论
通过提交评论,您同意遵守我们的条款和社区准则。如果你发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。