摘要
突变在癌症的发展中起着至关重要的作用,许多突变还会产生可靶向的脆弱性。确定含有突变基因的人群中所有癌症病例的比例,对公共卫生和基础科学都有好处。在这里,我们通过结合基因组和流行病学数据提供了第一个这样的估计。我们估计KRAS仅在11%的癌症中发生突变,低于PIK3CA(13%),略高于BRAF(8%)。TP53是最常见的突变基因(35%),KMT2C、KMT2D和ARID1A是十大最常见的突变驱动基因,突出了表观遗传失调在癌症中的作用。主要癌症亚分类的分析强调了对个体癌症驱动因素的不同依赖。总的来说,我们发现,与局限于癌症类型子集的研究相比,癌症遗传学较少受到高频、高知名度的癌症驱动基因的支配。
类似的内容被其他人浏览
介绍
人们早就认识到,对癌症患者的基因组进行测序将确定那些与人类健康和疾病有关的基因。来自癌症患者肿瘤样本的DNA基因组测序现在已经表征了所有基因和许多不同形式癌症的相对突变丰度2,3,4,5,6,7。鉴定常见突变基因对药物开发非常重要,因为一些癌蛋白已被证明是有价值的药物靶标8,9,10,11,12,13,14。
更好地了解哪些基因在所有癌症中最常发生突变,以及以何种频率发生突变,可以帮助确定基因和途径的优先顺序,从而增加公共卫生效益。例如,人们普遍认为RAS癌基因在30%的人类癌症中发生突变。美国国家癌症研究所(National Cancer Institute)创建了Ras倡议,将重点放在Ras上,因为人们认为Ras突变癌症患者的负担很高,而且尚未得到满足。沿着这些思路,更好地了解来自已知癌症驱动因素的负担可能有助于更好地优先考虑研究资金。
对癌症患者中常见的不同突变的更好理解也有助于药物开发和个性化医疗。临床试验越来越多地采用一揽子试验的形式,即用一种蛋白质的突变形式对不同类型的癌症进行等效治疗。例如,larorectinib和entrectinib都获得了FDA的批准,部分原因是基于篮子试验数据显示对多种肿瘤类型的疗效。此外,pembrolizumab还获得了基于篮式试验数据的FDA批准17,20,21。由于FDA的批准可能越来越多地基于癌症类型未知的突变18,20,因此发现在不同组织中共同存在的突变的好处增加了。个体化医疗也在取得进展,通过fda批准的药物在标签外的使用,试图抑制除当前批准的22、23、24、25中指定的癌症形式的靶向变异。改进的靶基因突变总体流行率的特征可能有助于设计这种个性化医学临床试验。
虽然携带基因突变的患者肿瘤样本的比例可以很容易地用于许多特定类型的癌症,也可以从多个结合了各种癌症样本的泛癌症研究中获得26,27,28,但该领域尚未对人群中携带特定基因突变形式的癌症患者的总体百分比进行估计。乍一看,所有癌症中准确的突变患病率值都是不可获得的,这可能令人惊讶,因为这些信息似乎很容易从诸如cbioportal29,30, COSMIC31, tcga26,27, AACR GENIE32和MSKCC impact等资源中获得。虽然这些资源中的一些可以提供给定基因突变的样本百分比的值,但这些资源中不同癌症的表示并没有被设计成与人群中这些癌症的相对负担成正比。
如果用每种癌症的相对丰度对每种癌症中每种基因的突变频率进行加权,就有可能计算出具有基因突变的人群中癌症的总体比例。(注:我们将使用术语突变比例来表示观察到癌症患者肿瘤内突变的总体水平概率。)美国国家癌症研究所(NCI)监测、流行病学和最终结果(SEER)项目自20世纪70年代中期以来一直追踪美国的癌症发病率。这些数据被用于美国癌症协会报告的年度癌症流行病学调查。然而,由于基因组学和流行病学对癌症分类系统的使用缺乏一致性(图1),不可能通过流行病学观察到的相同癌症的病例数来简单地加权癌症基因组学研究中的突变频率。SEER通过两个国际肿瘤疾病分类第三版(ICD-O-3)代码36来表征每种癌症。一种指定了起源部位的解剖位置,另一种指定了肿瘤的组织学,许多癌症需要这两种方法进行唯一的分类。相比之下,许多癌症基因组学研究并不为其样本提供ICD-O-3编码,而是用宽泛的通用术语描述其病理诊断,如乳腺癌或胰腺腺癌。
在这里,我们计算并展示了在基因组的每个基因中包含一个或多个突变的所有癌症的估计比例。我们通过整合流行病学和基因组数据来做到这一点。我们通过开发和实现这两种数据类型之间的映射过程,克服了癌症分类系统缺乏一致性所造成的障碍。我们的研究结果表明,特定的癌症驱动突变比人们认为的要少得多,一些引人注目的致癌基因比通常所说的要少得多。
结果
罗塞塔的发展
为了整合癌症基因组数据和SEER流行病学数据,我们开发了一种方法来相互转换用于标记癌症诊断的不同命名法。我们通过创建ROSETTA(重新分类测序和流行病学肿瘤类型注释)来做到这一点。ROSETTA实际上是一个可替代的分类系统,它映射到其他两个分类系统,因此它能够整合流行病学和基因组数据(补充文件1;ROSETTA分类是通过将类似的ICD-O-3分类分组在一个类别中创建的,就像癌症基因组学研究可能专注于一种一般形式的癌症(即肺腺癌),其中收集来自流行病学记录中使用的25种或更多详细的ICD-O-3腺癌亚型的样本池一样。
我们的ROSETTA分类既有生物学的影响,也有实用主义的影响。分组是根据已经纳入ICD-O-3代码整体组织的生物因素进行的。分组的粒度也受到先前癌症测序研究中所做选择的影响。例如,当相应的测序研究非常广泛,无法根据元数据进行细分时,ROSETTA分类就会广泛地映射到SEER分类上。当基因组研究可以在纳入元数据的基础上进一步细分时,可以为单个基因组研究使用多个ROSETTA分类术语,以提高数据集之间映射的分辨率。我们的方法产生了370种不同粒度水平的不同癌症分类类别。在ROSETTA中癌症细分的精细程度的混合粒度表明,ROSETTA分类的数量不应被用作任何一个细节水平上癌症类型数量的度量,而应被视为该工具规模的度量,该工具能够实现基因组和流行病学的整合。
ROSETTA对流行病学数据的重新分类
我们利用SEER流行病学数据跟踪2000年至2017年间的恶性癌症诊断,其区域分布覆盖了约25%的美国人口34(图2a)。我们认为在这个人群中诊断出的癌症比例是对整个美国人口中诊断出的癌症比例的合理估计;支持我们断言的是,美国癌症学会每年对全国癌症发病率的流行病学估计35与这些数据非常相关,Pearson的r = 0.98和p < 0.00001(图S1)。总体而言,该SEER数据集包含超过700万种不同的癌症诊断,每种诊断由670种ICD-O-3恶性癌症组织学(形态学)代码中的一种和100多种ICD-O-3位点(解剖位置或地形)代码中的一种指定(图2a)。我们根据ROSETTA对流行病学研究的组织学诊断进行重新分类,并在不同的处理间隔进行质量控制检查,以确保数据处理的有效性(图S2)。
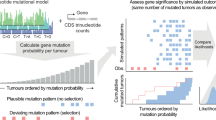
a我们通过实施ROSETTA癌症分类实现癌症基因组和流行病学数据的整合。b比较以往TCGA泛癌症分析和本研究中具有代表性基因组测序数据的所有流行病学癌症发病率的比例。c TCGA泛癌症数据集中属于特定癌症类型的比例与相同癌症类型的流行病学比例的比较,以及Pearson相关性和p值(双尾)(左面板p = 0.07,右面板p < 0.00001)。d本研究的加权基因突变比例与kandoes等人的突变发生率的比较。TCGA泛癌分析。e本研究的加权基因突变比例与Bailey等人的突变发生率的比较。TCGA泛癌分析。在面板c-e中,x = y对角线表示两个数据集之间的偏差。图d和e中的误差条表示95%置信区间,y坐标表示计算的突变比例,x坐标是来自文献的数据点,没有可用的误差估计。
在ROSETTA重新分类之后,我们找到了每种癌症的总发病率,并将这些值格式化为向量,该向量提供了每种ROSETTA类别中所有癌症的比例(补充数据1)。五种最常见的癌症类型是乳腺癌、前列腺癌、结直肠腺癌、肺腺癌和恶性黑色素瘤,它们总共占所有癌症诊断的50%以上。最常见的10种类型占所有癌症诊断的68.7%。值得注意的是,一些泛癌症研究并没有包括10种最常见的癌症类型的样本。
通过ROSETTA和汇总分析对基因组研究进行重新分类
对于癌症测序数据,我们纳入了来自139个不同癌症测序研究的19181名癌症患者的外显子组(图2a)。我们获得了以前在人类癌症患者样本上进行的基于外显子组的基因组研究的突变要求。所利用研究的完整清单见补充数据2。我们根据附加的元数据对测序样本进行手动整理和分配ROSETTA代码,这些元数据揭示了样本更精细的组织病理学(图S2)。然后,我们自动记录每个样本中至少有一次突变的基因,包括错义突变、无义突变或短indel,并在每个样本指定的ROSETTA分类中保持这些计数。由于我们的目标是将基因突变(或不突变)的癌症患者比例制成表格,我们将患者样本计算为突变,无论其在同一基因中有单个突变还是多个突变。当对同一患者的多个样本进行测序时,我们仅将原始样本纳入分析。此外,一些癌症基因组学研究将先前发表的样本与他们的新样本结合起来。为了避免重复计算相同的样本,我们在患者和样本识别分类器的基础上确定了研究之间的重叠样本,并且我们在分析中只包括来自最近研究的突变呼叫。
基因突变的方式多种多样,包括内含子和外显子区域的突变38,39。在我们目前的分析中,我们将重点放在改变基因编码的突变上,这些突变可以在外显子组数据中以高速率可靠地检测到,包括错义突变、无义突变和indel突变。我们强调,我们在本研究中使用的术语突变仅限于这些类型的突变,并且我们不包括基因融合,内含子(例如,剪接位点)突变或拷贝数突变(即扩增或缺失)。现在我们已经开发并提出了ROSETTA,我们将流行病学数据与基因组数据整合的方法可以在未来的研究中应用于其他类型的基因组数据和其他形式的突变。
癌症类型覆盖的评估及与以往泛癌症研究的比较
随着SEER和癌症基因组学根据ROSETTA重新分类,整合这些数据成为可能。我们估计癌症患者群体中任何基因突变的总体比例为一种癌症类型的基因突变比例与由该癌症类型引起的所有癌症发病率的百分比的乘积之和(图S2)。
尽管有许多不同类型的癌症,但更常见的癌症在所有癌症中所占的比例很大。我们发现我们的测序数据代表了93%的癌症病例(图2B)。在我们这里提供的估计中,我们考虑这93%的诊断中的流行病学加权突变比例,以近似于基因组测序数据不容易获得的剩余7%的癌症诊断的突变频率。我们注意到,其他泛癌症和汇总分析隐含着类似但更强的假设,这些分析不考虑癌症的流行病学发病率,并且包括较小比例的癌症类型。我们还注意到,对另外7%的癌症诊断进行进一步测序将消除对这一假设的需要,并将揭示这一假设是否对总体估计具有生物学意义的影响。
我们将加权突变比例与两个(未加权的)TCGA基因突变发生率泛癌分析进行了比较。kandos等人26进行的首次泛癌症TCGA分析获得了52%癌症病例的代表性测序数据(图2b)。每种癌症的样本数量与总体癌症频率不成比例(图2c)。我们比较了125个蛋白质编码基因的突变频率,kandoes等人的分析得出这些基因是癌症驱动基因,并提供了泛癌症突变频率26。尽管数据集中突变样本的比例与我们的流行病学加权估计之间存在良好的总体一致性,但对每项研究结果的比较表明,如果认为未加权的泛癌症分析代表一般癌症患者群体中的突变比例,则会犯错误。例如,未加权研究发现VHL和BRAF突变发生率分别为7%和2%,而我们的加权研究估计这些基因在1%和8%的癌症人群中发生突变。这些差异很可能源于TCGA泛癌症数据集没有被设计成与癌症发病率成正比,并且相对于其他癌症类型,具有这些突变的癌症分别被过度代表和低估(图2d)。
Bailey等人最近的TCGA泛癌症分析27具有85%癌症病例的代表性测序数据(图2b),每种癌症的测序样本数量更符合癌症诊断频率的比例(图2c)。我们比较了Bailey等人分析得出的299个基因的突变频率,这些基因是癌症驱动基因,也呈现了泛癌症突变频率27。尽管在未加权的泛癌症突变频率和我们的流行病学加权突变比例之间有很好的总体一致性,但许多个体基因的突变率在不同的方法之间存在差异。例如,这项未加权的研究发现KRAS、APC和IDH1的突变频率分别为7.6%和6%,而我们的加权研究发现它们的突变率分别为11.10%和1%(图2e)。因此,尽管Bailey等人的研究提高了癌症类型覆盖率,提高了相对比例代表性,但考虑这些值作为实际突变频率的估计,对于一些常见突变基因仍可能导致相对较大的误差。
值得注意的是,我们没有黄金标准来比较基于rosetta的估计。然而,随着癌症类型的覆盖范围和不同癌症的比例代表性的提高,泛癌症基因突变频率预计将是对真实频率的更好估计。在这些泛癌症研究和我们基于rosetta的估计之间可以看到这种趋势。这为我们的方法及其实现提供了一些额外的验证。
癌症发病率突变频率估计
我们将重点放在计算出的COSMIC癌症基因普查列表(Tier 1)中基因的种群水平突变比例上(图3a;表1;和补充数据3)TP53是最常见的突变癌症驱动基因,我们估计在35%的新癌症诊断中发生了突变。尽管KRAS被广泛认为是最常见的突变原癌基因40,41,42,但我们的流行病学加权估计发现,KRAS突变(11%)比PIK3CA突变(13%)更少见。研究发现,BRAF致癌基因在8%的癌症中发生突变,其发生率仅略低于KRAS。三个表观遗传修饰因子(KMT2C, KMT2D和ARID1A)是10个最常发生突变的癌症驱动基因之一,突出了癌症中表观遗传失调的频率43。总的来说,令人惊讶的是,最常发生突变的癌症相关基因仅在所有癌症的一小部分中发生了突变。由于我们的分析既包括驱动基因突变,也包括乘客基因突变,并且仅仅着眼于癌症基因普查中基因内发现的突变总数,这表明人类癌症中驱动基因突变的总体发生率甚至低于我们在这里提出的值。
具有致病性突变的蛋白激酶已被证明是有价值的药物靶点,多种fda批准的小分子抑制剂已被开发8,9,10,11,12,13,14,44。因此,我们考虑了所有蛋白激酶的总体突变频率(图3b,补充数据3)。我们发现两种最常见的突变激酶是TTN(30%)和obn(9%)。TTN和obcn编码两个最大的具有激酶结构域的蛋白质(分别约34,000和8,000个氨基酸),这些基因的大尺寸可能导致它们的高突变频率45。尽管可能存在一些理论上的争论,认为非常大的基因中的一小部分突变可能不是乘客突变,但这些激酶的突变通常被认为是乘客突变,这两个基因不包括在COSMIC癌症基因普查列表中。相比之下,BRAF和ATM都是公认的癌症驱动基因,这两个含激酶的基因分别在8%和5%的癌症中发生突变。据估计,所有其他蛋白激酶在所有癌症中发生突变的比例小于5%。与没有(或低)选择压力的大基因相比,BRAF和ATM等具有强促癌突变选择压力的激酶基因发生突变的几率要小得多,这可能表明,每个驱动基因的选择压力并非存在于每个组织中,并且/或者可能表明选择压力受到共同发生的突变的影响。再次,我们注意到我们计算的突变比例适用于所有突变,而不限于那些最有可能致病的突变。此外,重要的是要注意,并非驱动基因激酶的所有突变形式都是相同的靶标47。因此,这些值将高估在这些激酶中发现潜在可靶向致病性突变的频率。
我们还考虑了由NCI RAS initiative定义为RAS通路成员的基因。Ras通路在人类癌症中具有明确的作用,该通路的突变能够赋予多种癌症特征48,49。此外,科学文献通常估计30-33%的癌症在KRAS、NRAS或hras50,51,52中存在突变。相比之下,我们的综合基因组流行病学方法提供了15%的癌症携带KRAS, NRAS或HRAS突变的修订估计-不到先前报道估计的一半。我们关于RAS基因突变在所有癌症中所占比例的研究结果与最近一项对KRAS、NRAS和HRAS突变进行更简单加权分析的分析一致。这项研究还包括拷贝数变异,估计19%的癌症患者在这些RAS基因中有一个突变53。
正在进行的癌症驱动基因发现工作已经在RAS通路中发现了许多新的驱动因子27,54,55。就像我们对先前指定的癌症驱动因子和蛋白激酶的分析一样,我们对RAS通路基因突变的分析发现了少数常见突变基因,随后是一长尾不太频繁突变的基因(图3C;因此,尽管RAS通路中可能会有更多的基因被归类为癌症驱动基因,但似乎没有泛癌高患病率的RAS通路驱动基因有待发现。总之,我们的分析表明,癌症基因组学并不像个别癌症研究所暗示的那样,是由常见的、高频的驱动基因突变主导的。
泛腺癌和泛鳞状细胞癌的分析
癌症可以根据共同的组织学分为更高级别的组,如腺癌、淋巴瘤和胶质瘤。在ICD-O-3中,不同的组织学编码被组织在这样的高阶分类中36。我们的ROSETTA实现务实地在不同的高阶类别中采用不同的粒度级别,作为我们在可用测序数据的基础上做出准确的种群水平估计的努力的一部分。接下来,我们分析了这些高级分组水平上的测序数据,以评估基因突变比例模式在更一般的癌症分类之间的变化。
两种最常见的高级别癌症是腺癌和鳞状细胞癌(SCC)(图4a)。我们通过进行加权流行病学分析来计算泛腺癌和泛SCC突变比例,但仅限于所有ROSETTA腺癌(图4b,补充数据4)和所有ROSETTA SCC类别(图4c,补充数据4)。我们考虑了COSMIC癌症基因普查(Tier 1)突变在所有类型的腺癌(图4d)和所有形式的恶性鳞状细胞癌(图4e)的流行病学加权比例。我们还直接比较了这两种主要癌症亚型之间的突变率(图4f)。大多数基因在腺癌和鳞状细胞癌中的突变频率相差在5%以内。在腺癌中,仅有两个基因的净突变率高于5%,它们是KRAS和APC。据估计KRAS在14%的腺癌中发生突变,但在SCC中只有1%发生突变,而APC在13%的腺癌和5%的SCC中发生突变。7个基因倾向于SCC,总体突变率比腺癌高5%。据估计,在63%的鳞状细胞癌和34%的腺癌中,TP53发生了突变,这使得TP53成为两种主要癌症分类中最常见的突变癌症基因。其他在SCC中发生更多突变的基因(分别在SCC和腺癌中估计突变比例)是LRP1B (23%, 10%), KMT2D (16%, 7%), FAT1 (15%, 5%), CDKN2A (12%, 2%), NOTCH1(10%, 3%)和NFE2L2(10%和1%)。
a流行病学研究中属于腺癌(AC)、鳞状细胞癌(SCC)、恶性黑色素瘤(MM)或移行细胞癌(TCC)等主要亚分类的所有癌症的比例。b交流设备的具体类型及其在所有交流设备中所占的相对比例。c恶性鳞状细胞癌的具体类型及其在所有鳞状细胞癌中的相对比例。d流行病学加权泛ac分析前25个突变基因。e流行病学加权泛scc分析前25个突变基因。f癌症基因普查基因在AC和SCC之间的突变比例比较。选择95%置信区间不重叠的基因。所有基因及其突变比例和置信区间列于补充数据4。g流行病学加权泛黑色素瘤分析前25个突变基因。h TCC前25个突变基因。I KRAS、NRAS和HRAS发生突变的每种主要癌症亚型的比例。所有图d、e和g-i中的误差条表示通过模拟样本确定的95%置信区间(n = 2,000个独立泊松分布计算样本,每个基因和组织以计算的突变比例为中心值),条形水平表示计算的突变比例。
接下来的两个主要组的癌症在我们罗塞塔子分类ICD-O-3类别是黑色素瘤和移行细胞癌(补充数据4)。我们为每一个下一个评估突变比例最丰富的子分类(图4 g h)。最常BRAF基因变异的黑色素瘤,使黑色素瘤唯一的主要类别的癌症TP53不是最常见的突变基因和PIK3CA不是最常见的致癌基因。此外,移行细胞癌的10个最常见突变基因中有7个直接参与表观遗传和转录因子调控。
我们还考虑了三种RAS基因(KRAS, NRAS和HRAS)在这四种实体瘤中的突变比例。我们发现,在至少一种主要的癌症类型中,这三种基因中的每一种都是突变最多的(图4i)。KRAS是腺癌中最常见的突变,而NRAS是黑色素瘤中最常见的突变。有趣的是,在鳞状细胞癌和移行细胞癌中,HRAS是最常见的RAS GTPase突变。KRAS通常被认为是促进癌症的三种RAS GTPases中最差的;我们的泛癌症分析表明,哪种致癌RAS GTPase最能促进癌症的问题可能取决于癌症发生的细胞类型。
讨论
总的来说,这项工作描述了我们整合流行病学和基因组数据的方法,为每个基因中携带编码外显子突变的美国癌症患者的总体比例提供真实世界的估计。我们关注的是癌症发病率的数据,定义为一段时间内的新病例。流行病学数据还包括患病率(在特定时间内患有某种疾病的人数)和死亡率(在一段时间内因癌症死亡的人数)。我们选择不计算死亡率和患病率的流行病学加权估计,因为临床结果可能取决于癌症中存在的特定基因突变56,57,58。在这里,我们假设被测序的可能性更多地是发病率的函数,而不是存活率的函数。然而,对哪些肿瘤进行测序的决定可能受到其他因素的影响,如肿瘤大小和分期。这些选择和实践超出了当前工作的范围,可能会在基础数据中引入偏差,然后在计算的突变比例估计中反映出来。随着ROSETTA框架的建立,应该可以将这种流行病学加权方法应用于其他类型的测序数据,以调查拷贝数改变、内含子突变39、基因融合59和甲基化模式60。
我们强调,这里提出的基因突变比例,尽管是根据高质量的流行病学和基因组数据计算出来的,但仍应被视为估计值。癌症基因组研究不一定能准确反映人群中患有特定类型癌症的所有个体,无论是由于种族、民族和/或地理差异,还是由于接触致癌物和发病年龄的差异。此外,主要癌症类别的更精细的组织学细分可能无法在给定形式的癌症的整个肿瘤样本集合中按比例表示。我们的ROSETTA地图并不是整合流行病学和基因组数据的唯一可行方法,其他人也可以采用与这里相同的一般策略,但做出不同的选择。我们预计,随着癌症基因组学变得越来越普遍,随着测序样本的注释越来越好,随着罕见癌症的测序越来越多,估计将继续提高。在某种程度上,肿瘤测序可能非常普遍,甚至不需要采样,因为人群中的所有癌症都将在初始检测时接受测序。这种情况将克服根据样本进行推断以计算估计的需要。它将克服对尚未测序的癌症类型的基因进行假设的需要。此外,这种情况也将克服数据中可能存在的偏差,比如如何以及何时选择肿瘤作为一种癌症的代表性样本并将其发送给测序。在所有患者的肿瘤测序完成之前,我们相信,对于那些希望了解他们感兴趣的基因在癌症中突变的普遍程度的癌症研究人员来说,这里提供的值是有用的估计。
RAS突变通常在30-33%的癌症患者中发现50,51,52。我们已经表明,RAS突变在所有癌症中约占15%,这支持了之前非流行病学加权泛癌症分析中观察到的趋势26,27,33,并且与最近更简单的流行病学加权RAS突变估计一致53。考虑到两种最常见的癌症,乳腺癌和前列腺癌,占美国所有新诊断癌症的29%,很少有RAS基因突变,历史估计和最近估计之间的差异是合理的。此外,RAS突变发生率最高的相对常见的癌症是胰腺腺癌,在近90%的测序样本中都有KRAS突变。然而,胰腺腺癌对人群水平估计的影响是有限的,因为在给定年份中,小于3%的新癌症诊断是胰腺腺癌34,35。因此,对RAS突变流行率的常见估计似乎未能解释这样一个事实,即通常含有RAS突变的癌症不如很少有RAS突变的癌症常见。准确估计RAS基因以及其他基因的重要性,可以从最近两项备受瞩目的高成本研究工作中推断出来:NCI RAS计划和DARPA大机制计划52,63。与其他致癌基因相比,RAS突变的高丰度在一定程度上证明了这一点。因此,我们提出的准确流行病学估计可能会影响未来致力于关注癌症常见突变驱动因素的努力。
KRAS突变率低于预期也表明,纵观所有癌症病例,人类癌症中几乎没有高频突变。这对药物开发具有启示意义,其中针对常见致癌突变的小分子靶向治疗已成为主要焦点。尽管这些药物显然对许多人有益,但这一分析表明,所有癌症患者从任何一种针对特定突变基因产物的靶向治疗中受益的比例将是有限的。高频泛癌症驱动因素的惊人缺乏也对癌症的发展具有重要意义,因为它表明癌症可能不像特定癌症研究所暗示的那样由高频驱动因素主导。这进一步表明,本文未考虑的其他因素,如拷贝数变异、表观遗传基因突变以外的表观遗传失调、融合蛋白和微环境线索,都可能在趋同性癌症表型中发挥重要作用48,49。识别趋同表型及其可靶向脆弱性的方法可能比仅关注特定突变的努力提供更好的机会使许多患者受益64,65。
方法
肿瘤组织学再分类
我们开发了ICD-O-3形态学(组织学)和地形(解剖位置)编码对之间的映射,以及癌症基因组学中使用的肿瘤分类描述符之间的映射,我们将其称为ROSETTA(测序和流行病学肿瘤类型注释的重新分类)。为此,两位接受过病理学、癌症生物学和生物信息学培训的医生系统地、反复地检查了ICD-O-3和癌症基因组注释文件,以评估需要哪些级别的癌症分组来解析数据集。开发ROSETTA分类是为了作为一种常见的癌症分类系统,流行病学和基因组癌症分类器可以在其上进行映射。然后,我们在重新分类的基础上,将ICD-O-3代码中的地图指定给ROSETTA(补充文件1)。此外,我们在患者元数据的基础上开发了从癌症患者文件到ROSETTA的地图。制定ROSETTA分类是为了与ICD-O-3的主要组织原则保持一致。
流行病学数据和处理
癌症流行病学数据采用SEER*Stat软件从NCI监测研究计划数据库SEER中获取。我们使用的数据库名为:Incidence - SEER Research Data, 18 registers, Nov 2019 Sub(2000-2017)。我们在表输出中导出恶性诊断数据,其中行由ICD-O-3历史/行为代码指定,列由站点代码ICD-O-3/WHO 2008位置代码指定。
处理有多个步骤(补充文件1)。简单地说,步骤包括使用ACS在其年度癌症流行病学报告中使用的分辨率将其分组为更高级别的解剖位置位点代码35,处理组织学和起源位置不明确的样本,以及通过ROSETTA进行重新分类。在每个处理步骤之后,我们都会检查研究中的样本总数是否恒定,以确保在我们的实施过程中不会丢失数据。我们处理的SEER数据的输出是一个表格,其中行为43个位点(解剖位置),列为370个ROSETTA分类。表中的值是该部位每种组织学的估计计数。
癌症基因组数据
我们利用了公开的,非禁运的,非临时的癌症基因组学研究,这些研究被包括在cbioportal29,30上。我们纳入了组织样本的外显子组测序研究,即,不是来自异种移植物或细胞系(补充数据2)。我们专注于无义和错义突变,以及小的索引。在cBioPortal符号中,如果患者体内的基因有以下一个(或多个)突变,我们将其视为突变:nonsense_mutation、frame_shift_del、frame_shift、frame_shift_ins、missense_mutation、missense、nonsense、in_frame_del、in_frame_ins、nonstop_mutation。我们没有包括剪接位点或融合突变。我们用适当的ROSETTA注释重新注释每个患者样本的突变注释格式(MAF)文件。我们的基因组处理步骤的输出是一个表格,其中列出了每个基因突变的样本总数,为每个ROSETTA癌症分类完成。当一项研究包括纵向样本(即,同一患者多次采样)时,我们只使用第一个样本。当多个研究包括来自同一患者样本的数据时,我们使用了来自最近研究的突变数据。由于同一基因可能有不同的名称,我们还使用cBioPortal文档中描述的人类基因名称图对基因名称进行了标准化。所使用的癌症基因组数据来自各种来源,包括TCGA和TARGET计划。这里公布的结果部分基于TCGA研究网络(https://www.cancer.gov/tcga)和产生有效治疗的治疗应用研究(https://ocg.cancer.gov/programs/target)计划(phs000218: https://portal.gdc.cancer.gov/projects)产生的数据。在补充数据2的表格中提供了所有基因组学研究的列表。该表还列出了在每个基因组学研究中分配给一个或多个样本的ROSETTA代码。从每个样本映射到ROSETTA代码的文本文件可在补充软件中获得。有关绘图和软件实施的其他信息见“补充方法”。
突变比例估计
我们将我们的基因组输出表转换为一个表,该表列出了在ROSETTA分类中含有给定基因突变的已测序肿瘤的观察比例。换句话说,我们将基因组输出表转换为m × n矩阵(C),其中m是基因的数量,n是已测序的ROSETTA分类的数量。在我们目前的研究中,m的值为21,271,n的值为73。C中的值表示给定ROSETTA分类的基因突变的条件概率。
我们将SEER流行病学输出转换为列出每个ROSETTA代码中所有代表性癌症的数量。这导致了一个k × 1矩阵S,其中k是所有ROSETTA分类的数量(370),k > n,因为不是所有ROSETTA分类都有代表性的测序数据。我们有ROSETTA代码的代表性测序数据,占所有观察到的人类癌症的93%。我们假设93%的癌症的突变加权比例是对所有癌症加权比例的一个很好的估计。因此,我们通过消除ROSETTA分类中没有代表性基因组测序数据的所有行,将S转换为S',这是一个n × 1矩阵。我们标准化了这个向量,所以它的和是100%。
然后,可以通过将ROSETTA分类中的突变率与所有ROSETTA分类中具有该ROSETTA分类的所有癌症的比例相加来计算估计的突变比例。或者,C•S = G,其中G是一个m × 1矩阵,列出了所有基因的总体加权突变频率(即图1,底部)。
我们通过构建癌症突变观察的计算机研究,对基因组数据集进行了统计分析。由于在任何给定的组织或组织学中,典型的基因组研究(总共19,181个样本)比流行病学研究(总共7,167,808个样本)小两个或两个以上的数量级,因此我们将基因组研究作为我们结果中任何差异的主要来源。我们进行了统计分析,假设泊松分布适用于研究期间观察到的癌症病例。我们为基因和组织学的每一种组合生成2000个泊松分布样本,其中包括从基因组研究中计算出的中心值。通过我们的重新加权(条件概率)管道(图S2)处理每个硅样品,计算每个基因突变病例比例的重复(图S2),并从这些重复中计算95%的置信区间。
报告总结
关于研究设计的更多信息可以在本文链接的《自然》研究报告摘要中找到。
数据可用性
本研究未产生新数据。输入的基因组数据可从www.cbioportal.org公开获得,所有包含的基因组研究的url在文档(补充数据2)中作为表格列出。输入的流行病学数据可通过SEER- stat程序公开下载,该程序可在NCI SEER注册后通过网站https://seer.cancer.gov/seerstat/下载。复制这项工作所需的所有基因组数据都可以在补充软件1中的压缩文件Genomics_Analysis\interim_files\df_mutfiles.zip中以ROSETTA重新标记(处理)的基因组数据的形式获得。复制这项工作所需的所有流行病学数据都可以在补充软件1中的文件SEER_Analysis/Output_SEER.txt中以ROSETTA重新标记(处理)的流行病学数据的形式获得。稿件中描述的所有输出数据均在“补充信息/补充数据”中提供。
代码的可用性
用于结合基因组学和流行病学数据并生成手稿中所有图表和表格的源代码作为补充压缩文件夹呈现,也可以从链接https://github.com/GMendiratta/ROSETTA-for-Cancer-Mutations下载。下载和处理基因组数据的源代码以及处理原始SEER数据的源代码也分别包含在名为Genomics_Analysis/和SEER_Analysis的文件夹中。代码是使用Jupyter notebook IDE用python 3编写的,并使用NumPy、pandas、random和Matplotlib库。此代码是按原样提供的,可以复制、重用和编辑,并注明本文的引用。不需要作者的额外许可。
参考文献
癌症研究的转折点:人类基因组测序。科学231,1055-1056(1986)。
癌症基因组图谱研究网络。全面的基因组特征定义了人类胶质母细胞瘤基因和核心途径。自然,455,1061-1068(2008)。
癌症基因组图谱网络。人结肠癌和直肠癌的综合分子表征。《自然》,2012,33 - 37。
癌症基因组图谱研究网络。肺腺癌的综合分子谱分析。自然511,543-550(2014)。
Biankin, a.v.等人。胰腺癌基因组揭示轴突引导通路基因畸变。《自然》(英文版),2012。
霍迪斯,E.等。黑色素瘤的驱动突变。Cell 150,251 - 263(2012)。
癌症基因组图谱研究网络。子宫内膜癌的综合基因组特征。《自然》(英文版),2013年第7期。et al。
博拉格,G.等。一种RAF抑制剂在braf突变型黑色素瘤的临床疗效需要广泛的靶点阻断。自然,467,596-599(2010)。
莫承祥等。奥西替尼或铂-培美曲塞治疗EGFR t790m阳性肺癌心血管病。[j] .医学杂志,2016,36 (2);
查普曼,p.b.等人。vemurafenib提高BRAF V600E突变黑色素瘤患者的生存率。心血管病。[j] .医学杂志,2004,17(4):444 - 444。
海因里希,m.c.等。转移性胃肠道间质瘤患者的激酶突变和伊马替尼反应。j .中国。中国医学杂志21,4342-4349(2003)。
Perl, a.e.等。吉替尼或化疗治疗复发或难治性flt3突变AML。心血管病。中华医学杂志,2003,19(2):444 - 444。
马,c.x.等。Neratinib疗效和循环肿瘤DNA检测在HER2非扩增转移性乳腺癌中的HER2突变。中国。癌症杂志23,5687-5695(2017)。
Loriot, Y.等。厄达非替尼治疗局部晚期或转移性尿路上皮癌。心血管病。中华医学杂志,2016,38(2):444 - 444。
美国国家癌症研究所的新Ras项目瞄准了一个老敌人。新医学19,949-950(2013)。
redg, A. J. & Janne, P. A.篮子试验和基因组医学时代临床试验设计的演变。j .中国。中华医学杂志。33,975-977(2015)。
Park, J. J. H, Hsu, G., Siden, E. G, Thorlund, K. & Mills, E. J.对临床医生的精准肿瘤篮子和伞式试验的概述。[j] .中国临床医学杂志,2016,32(2):481 - 481(2020)。
FDA批准具有里程碑意义的组织不可知论癌症药物。Nat Rev. Drug Disco. 18,7(2018)。
Drilon等人。larorectinib治疗成人和儿童TRK融合阳性癌症的疗效。心血管病。中国医学杂志,2018,32(4):444 - 444。
Marcus, L., Lemery, S. J, Keegan, P. & Pazdur, R. FDA批准总结:pembrolizumab用于治疗微卫星不稳定性高的实体瘤。中国。巨蟹座,25,3753-3758(2019)。
Le, d.t.等人。PD-1在错配修复缺陷肿瘤中的阻断作用。心血管病。中华医学杂志,2003,26(2):559 - 559(2015)。
Sicklick, j.k.等。I-PREDICT研究:癌症患者分子谱分析使个性化联合治疗成为可能。新医学25,744-750(2019)。
加藤,S.等人。来自分子肿瘤板的实际数据表明,精确的n -of- 1策略改善了结果。Nat. common . 11, 4965(2020)。
迪特里希等人。BRAF抑制难治性毛细胞白血病。心血管病。中国医学杂志,2003,26(4):444 - 444。
Haroche, J.等。vemurafenib治疗BRAF V600E突变的多系统难治性厄德海姆-切斯特病和朗格汉斯细胞组织细胞增多症的显著疗效[j] .中国科学:自然科学,2013。
坎多斯,C.等。12种主要癌症类型的突变景观和意义。《自然》(英文版),2013年第4期。
贝利,m.h.等人。癌症驱动基因和突变的综合表征。Cell 174, 1034-1035(2018)。
ICGC/TCGA全基因组泛癌分析联盟。全基因组泛癌分析。《自然》(英文版),2014,32 - 39。
陶瓷,等。cBio癌症基因组门户:一个探索多维癌症基因组数据的开放平台。《癌症迪斯科》,2012,41(2012)。
Gao, J.等。综合分析复杂的癌症基因组学和临床档案使用cbiopportal。科学。信号6,pl1(2013)。
桑卡,Z.等。宇宙癌症基因普查:描述所有人类癌症的遗传功能障碍。中华癌症杂志,18(6):696-705(2018)。
AACR项目精灵联盟。AACR GENIE项目:通过国际联盟推动精准医疗。巨蟹座迪斯科,7,818-831(2017)。
Zehir, A.等。1万名患者的前瞻性临床测序揭示了转移性癌症的突变景观。中华医学杂志23,703-713(2017)。
Hayat, M. J, Howlader, N., Reichman, M. E.和Edwards, B. K.来自监测、流行病学和最终结果(SEER)项目的癌症统计、趋势和多种原发癌症分析。肿瘤学家12,20-37(2007)。
西格尔,r.l.,米勒,k.d., Jemal, A.癌症统计,2020。中国临床医学杂志,2016,32(1)。
弗里茨a.p.等。国际肿瘤疾病分类第三版(世卫组织,2013)。
Leiserson, m.d.等。泛癌症网络分析确定了跨途径和蛋白质复合物的罕见体细胞突变的组合。中国生物医学工程学报,2015,32(2):444 - 444。
Watson, i.r., Takahashi, K., Futreal, p.a.和Chin, L.癌症体细胞突变的新模式。中国生物医学工程学报,2014,33(2)。
Diederichs等人。癌症基因组的暗物质:调控元件、未翻译区域、剪接位点、非编码RNA和同义突变的畸变。EMBO医学杂志,8,442-457(2016)。
斯蒂芬,a.g.,埃斯波西托,D,巴格尼,r.k.和麦考密克,f。中国生物医学工程学报(英文版),2014(6)。
林德赛,C. R.; Blackhall, F. H.。Br。[j] .中国医学杂志,2016,32(4):444 - 444。
Moore A. R, Rosenberg S. C, McCormick F., Malek S. ras靶向治疗:不可药物治疗吗?新药物发现,19,533-552(2020)。
黄文华,李建平,李建平,等。表观遗传可塑性与癌症的特征。Science https://doi.org/10.1126/science.aal2380(2017)。
Manning, G., Whyte, D. B., Martinez, R., Hunter, T.和Sudarsanam, S.人类基因组的蛋白激酶补体。科学298,1912-1934(2002)。
劳伦斯,m.s.等。癌症的突变异质性和寻找新的癌症相关基因。《自然》(英文版),2013年第4期。
格林曼,C.等。人类癌症基因组的体细胞突变模式。自然,446,153-158(2007)。
Yao, Z.等。具有3类BRAF突变体的肿瘤对活化RAS的抑制敏感。自然,548,234-238(2017)。
哈纳汉博士和温伯格博士。癌症的特征。Cell 100,57 - 70(2000)。
哈纳汉,D.和温伯格,R. A.。癌症的特征:下一代。Cell 144, 646-674(2011)。
Li, S, Balmain, A.和Counter, C. M.癌症中RAS突变模式的模型:寻找最佳位点。中华癌症杂志,18,767-777(2018)。
Keeton, a.b., Salter, e.a.和Piazza, g.a.。ras -效应相互作用作为药物靶点。中国癌症杂志,7,221-226(2017)。
你,J.人工智能。DARPA开始了。奥特曼。科学通报,2015(2)。
普瑞尔,胡德,F. E.和哈特利,J. L.癌症中Ras基因突变的频率。中国癌症杂志,20(5):649 - 649(2020)。
坎贝尔,j.d.等。肺腺癌和鳞状细胞癌中躯体基因组改变的不同模式。[j] .中国生物医学工程学报,2016,37(2):444 - 444。
霍恩,H.等人。NetSig:基于网络的癌症基因组发现。学报,15,61-66(2018)。
马克斯,j.l.等。EGFR和KRAS突变在切除肺腺癌中的预后和治疗意义。j . Thorac。中华医学杂志,3,111-116(2008)。
邢,M.等。BRAF突变预示着甲状腺乳头状癌较差的临床预后。j .中国。性。中国生物医学工程学报,2009,32(2):444 - 444(2005)。
Silwal-Pandit, L.等。乳腺癌中的TP53突变谱是亚型特异性的,具有明显的预后相关性。中国。中国癌症杂志,2014,35(5):379 - 379。
Stransky, N., Cerami, E., Schalm, S., Kim, J. L.和Lengauer, C.癌症中激酶融合的景观。学报,5,4846(2014)。
赵绍国等。晚期前列腺癌的DNA甲基化图景。中国生物医学工程学报,32(2),378 - 389(2020)。
斯普拉特博士等人。基因组测序中的种族/民族差异。中国生物医学工程学报,2016,32(2):444 - 444。
唐宁,j.r.等。儿童癌症基因组计划。植物学报,44,619-622(2012)。
DARPA的大型机械项目。理论物理。中国生物医学工程学报,2015,32(4):481 - 481。
金,j.w.等。分解致癌转录特征以生成不同细胞状态的图谱。细胞系统,5,105-118(2017)。e109。
Califano, A.和Alvarez, M. J.肿瘤起始、进展和药物敏感性的复发性结构。中华癌症杂志,17(5),391 - 391(2017)。
致谢
作者感谢Hannah Carter、Olivier Harismendy、Jason Sicklick、Pablo Tamayo、Peter Salamon以及Stites实验室的成员们的有益对话和评论。这项工作得到了先锋基金博士后学者奖、NIH K22CA216318、NIH T32CA009370、NIH P30CA014195和黑色素瘤研究联盟青年研究者奖的支持。
作者信息
作者及单位
贡献
e.k., m.a., g.m.和E.C.S.设计了这个项目。M.A.和E.C.S.开发了流行病学和癌症测序数据之间的ROSETTA映射,以共享另一种癌症分类。M.A.和G.M.开发了将ROSETTA整合到流行病学和基因组数据集的计算过程。e.k. d.l.和G.M.收集并处理了癌症基因组数据。g.m., m.a.和E.C.S.分析了流行病学数据。通用整合了流行病学数据和基因组数据。通用和E.C.S.进行了统计分析。D.L.和M.T.协助编写测试代码、验证和文档。E.C.S.和通用公司根据其他作者的意见撰写了这份手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益冲突。
额外的信息
《自然通讯》感谢Nicholas Zaorsky和其他匿名审稿人对这项工作的同行评审所做的贡献。同行评审报告是可用的。
施普林格·自然对已出版地图的管辖权要求和机构关系保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接从版权所有者处获得许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/。
关于本文
引用本文
Mendiratta, G., Ke, E., Aziz, M.等。美国人口的癌症基因突变频率。学报12,5961(2021)。https://doi.org/10.1038/s41467-021-26213-y
收稿日期:2020年11月24日
录用日期:2021年9月20日
发布日期:2021年10月13日
DOI: https://doi.org/10.1038/s41467 - 021 - 26213 - y
这篇文章是由
-
胚胎干细胞分化过程中MLL3/4去耦增强子H3K4单甲基化、H3K27乙酰化和基因激活的缺失
基因组生物学(2023)
-
Mdwgan-gp:基于多重鉴别器WGAN-GP的基因表达数据增强
BMC Bioinformatics (2023)
-
靶向USP2调控vprbp介导的p53和PD-L1降解用于癌症治疗
自然通讯(2023)
-
歌舞伎综合征患者发生的胚胎性横纹肌肉瘤的分子特征:根据肿瘤易感综合征的报告和文献综述
家族性癌症(2023年)
-
儿童和成人PTEN错构瘤肿瘤综合征患者的甲状腺表现:回顾性分析和文献复习
内分泌(2023)
评论
通过提交评论,您同意遵守我们的条款和社区准则。如果你发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。